 Sarà velocissimo e permetterà di localizzare con esattezza i nucleosomi, sorta di rocchetti proteici attorno a cui si avvolge quel lungo filo che è il nostro DNA: si tratta del Multi Layer Model, metodo computazionale sviluppato all’Università di Palermo da Vito Di Gesù del Gruppo di Bioinformatica del Dipartimento di Matematica e Applicazioni, in collaborazione con Davide Corona, ricercatore dell’Istituto Telethon Dulbecco che lavora grazie ai finanziamenti di Telethon e della Fondazione Giovanni Armenise-Harvard.
Sarà velocissimo e permetterà di localizzare con esattezza i nucleosomi, sorta di rocchetti proteici attorno a cui si avvolge quel lungo filo che è il nostro DNA: si tratta del Multi Layer Model, metodo computazionale sviluppato all’Università di Palermo da Vito Di Gesù del Gruppo di Bioinformatica del Dipartimento di Matematica e Applicazioni, in collaborazione con Davide Corona, ricercatore dell’Istituto Telethon Dulbecco che lavora grazie ai finanziamenti di Telethon e della Fondazione Giovanni Armenise-Harvard.
Questo studio interdisciplinare – pubblicato dalla rivista «Genomics» – può sembrare un esercizio di stile ai non addetti ai lavori. In realtà, identificare la posizione esatta dei nucleosomi nel nostro genoma potrebbe rivelarsi molto importante nello studio di numerose patologie umane (dal cancro a svariate patologie neurodegenerative) dovute a difetti nell’impacchettamento del DNA.
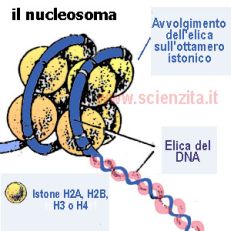
Infatti, come ricordato anche recentemente in un lavoro del citato Corona, il DNA presente nelle nostre cellule è avvolto attorno a dei rocchetti di proteine chiamati appunto nucleosomi, che si ripetono in modo regolare. Di fatto, si viene a formare una struttura che ricorda una collana di perle, dove il filo è il DNA e i nucleosomi le perle stesse.
Questa organizzazione è molto utile per due motivi. Innanzitutto permette di impacchettare in un piccolissimo nucleo di circa 6 micrometri di diametro (cioè 6 millesimi di millimetro!) una molecola che, se fosse srotolata, arriverebbe a circa due metri di lunghezza. Nello stesso tempo, l’impacchettamento del DNA su se stesso regola in maniera molto stretta quali geni debbano essere accessibili, e quindi “letti”, e quali invece debbano restare “silenziosi”.
Grazie al Multy Layer Model – realizzato anche con il contributo del Dipartimento di Biostatistica dell’Università americana di Harvard – si potrà calcolare la posizione dei nucleosomi con una velocità mille volte superiore a quella dei metodi attualmente usati.
Come spiega Vito Di Gesù, l’idea è scaturita guardando e riguardando i dati e dopo fitti dibattiti tra biologi e informatici sulla loro natura e grande variabilità.
«Alla base della metodologia che abbiamo sviluppato – spiega il ricercatore dell’Università di Palermo – c’è un concetto semplice: guarda il fenomeno da più punti di vista, scomponi il problema in sottoproblemi e determina i giusti collegamenti tra i vari frammenti di informazione. È un po’ come ricostruire un puzzle, ma in questo caso le tessere hanno i bordi sfumati. Nel nostro caso i diversi punti di vista sono la collezione dei tanti strati del segnale da analizzare, ottenuti a soglie diverse e secondo adeguati criteri di ottimizzazione. Ogni strato fornisce una tessera che contiene una parte dei segreti del puzzle».
(Ufficio Stampa Telethon)
Ufficio Stampa Telethon (Anna Maria Zaccheddu)
tel. 06 44015402, gcoppi@telethon.it.
Articoli Correlati
- Matematica, chimica, fisica: non più tabù per le persone con disabilità visiva «Una delle parole chiave che il mio staff ed io usiamo maggiormente è “tecnologia”, grazie alla quale, oggi, è possibile avere strumenti accessibili che consentono ai nostri studenti con disabilità…
- Sordocecità, la rivoluzione inclusiva delle donne Julia Brace, Laura Bridgman, Helen Keller, Sabina Santilli. E poi Anne Sullivan. Le prime quattro erano donne sordocieche, la quinta era “soltanto” quasi completamente cieca, ma non si può parlare…
- Ecco il meccanismo che “accende” il DNA Un gruppo di ricercatori dell’Istituto Telethon Dulbecco, attivo presso l’Università di Palermo, ha per la prima volta scoperto e osservato la molecola che, attivando i geni del DNA, permette alle…

